Spécifier un modèle linéaire pour comparer 2 groupes
Pour identifier les gènes différentiellement exprimés dans l’expérience sur la leucémie, vous devez traduire le modèle linéaire suivant en R :
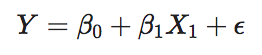
où \(X_{1}\) vaut 1 pour les cancers progressifs et 0 pour les cancers stables (remarque : R choisit automatiquement la condition de référence par ordre alphabétique).
Cet exercice fait partie du cours
Analyse d’expression différentielle avec limma en R
Instructions
L’objet ExpressionSet eset contenant les données de leucémie a été chargé dans votre environnement de travail.
- Utilisez
model.matrixpour construire une matrice de design avec un coefficient d’ordonnée à l’origine (interception) et un coefficient indiquant le statut de la maladie.
Exercice interactif pratique
Essayez cet exercice en complétant cet exemple de code.
# Create design matrix for leukemia study
design <- ___(~___, data = ___(eset))
# Count the number of samples modeled by each coefficient
colSums(design)