Créer l’objet DE
REMARQUE : le chargement de cet exercice peut prendre un peu plus de temps.
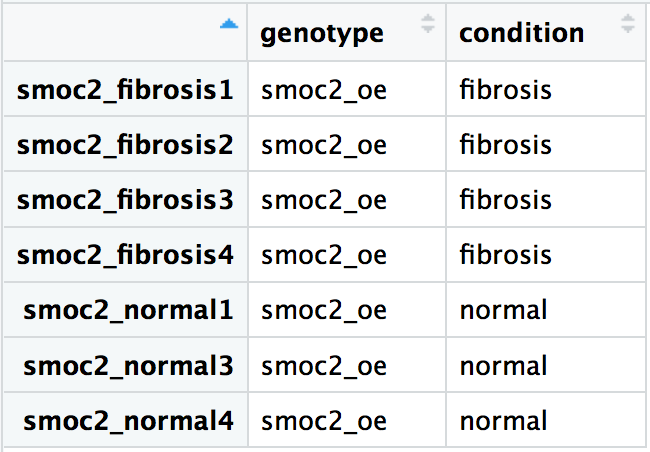
À partir de nos échantillons en surexpression de smoc2, créez l’objet DESeq2 de sorte que la formule de design spécifie la comparaison des différences d’expression entre les échantillons fibrose et normaux. Les métadonnées de l’expérience sont affichées ci-dessous. Nous avons lu les données avec les échantillons dans le même ordre pour les comptes bruts smoc2, reordered_smoc2_rawcounts, et pour les métadonnées, smoc2_metadata.
Cet exercice fait partie du cours
RNA-Seq avec Bioconductor en R
Instructions
Créez un objet DESeq2 appelé
dds_smoc2à l’aide de la fonctionDESeqDataSetFromMatrix()en spécifiant les arguments :countData,colDataetdesign.Exécutez la fonction
DESeq()pour estimer les facteurs de taille, calculer les dispersions et effectuer l’ajustement du modèle ainsi que les tests.
Exercice interactif pratique
Essayez cet exercice en complétant cet exemple de code.
# Create DESeq2 object
dds_smoc2 <- ___(___ = ___,
___ = ___,
___ = ~ condition)
# Run the DESeq2 analysis
___ <- ___(___)