DGE-Theorie: Metadaten
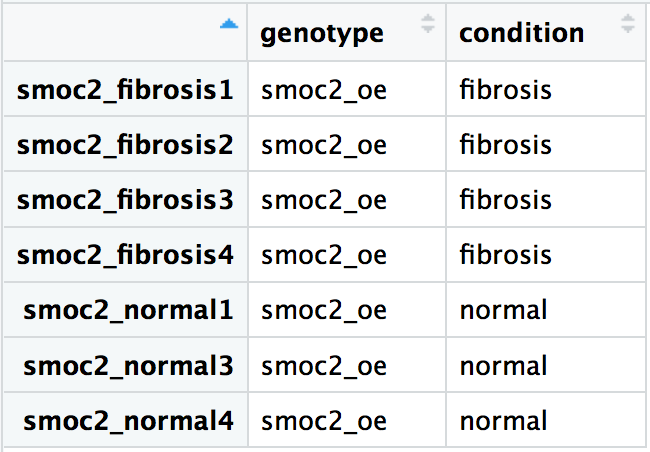
Verwende die Informationen unten, um für die Fibrose-Zähldaten einen Metadaten-Datenrahmen namens metadata mit den Spalten genotype und condition zu erstellen. Die Probennamen (z. B. smoc2_fibrosis1, smoc2_fibrosis2 usw.) sollen die Zeilennamen des Datenrahmens sein:
Diese Übung ist Teil des Kurses
RNA-Seq mit Bioconductor in R
Anleitung zur Übung
- Erstelle einen Zeichenkettenvektor namens
genotypefür die obigen Daten mitc(). - Erstelle einen Zeichenkettenvektor namens
conditionfür die obigen Daten mitc(). - Erstelle einen Datenrahmen namens
smoc2_metadatamitdata.frame()und den Zeichenkettenvektorengenotypeundcondition. - Erstelle einen Vektor mit Probennamen mittels
c()und weise ihn den Zeilennamen des Datenrahmens mitrownames()zu.
Interaktive Übung
Vervollständige den Beispielcode, um diese Übung erfolgreich abzuschließen.
# Create genotype vector
genotype <- c(___)
# Create condition vector
condition <- c(___)
# Create data frame
smoc2_metadata <- data.frame(___, ___)
# Assign the row names of the data frame
rownames(smoc2_metadata) <- c(___)