Erstellen des DE-Objekts
HINWEIS: Das Laden dieser Übung kann etwas länger dauern.
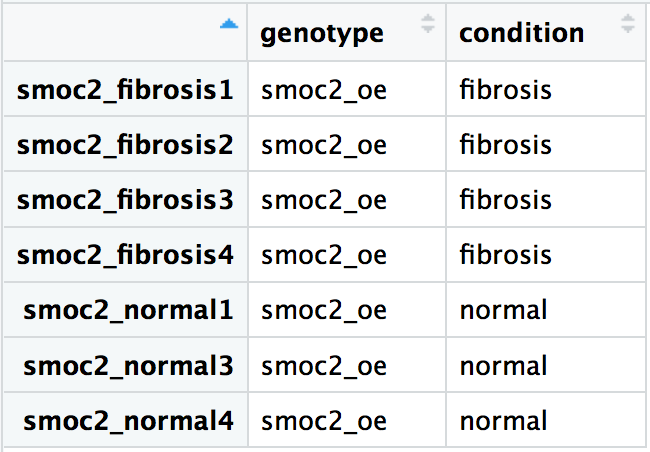
Nutze unsere smoc2-Überexpressionsproben und erstelle das DESeq2-Objekt so, dass die Design-Formel den Vergleich der Expressionsunterschiede zwischen Fibrose- und Normalproben angibt. Die Metadaten für das Experiment sind unten dargestellt. Wir haben die Daten so eingelesen, dass die Proben in derselben Reihenfolge vorliegen wie in den smoc2-Rohzählungen, reordered_smoc2_rawcounts, und in den Metadaten, smoc2_metadata.
Diese Übung ist Teil des Kurses
RNA-Seq mit Bioconductor in R
Anleitung zur Übung
Erstelle ein DESeq2-Objekt namens
dds_smoc2mit der FunktionDESeqDataSetFromMatrix()und gib die ArgumentecountData,colDataunddesignan.Führe die Funktion
DESeq()aus, um die Size-Faktoren zu schätzen, die Dispersionswerte zu berechnen sowie Modellanpassung und Tests durchzuführen.
Interaktive Übung
Vervollständige den Beispielcode, um diese Übung erfolgreich abzuschließen.
# Create DESeq2 object
dds_smoc2 <- ___(___ = ___,
___ = ___,
___ = ~ condition)
# Run the DESeq2 analysis
___ <- ___(___)