Teoria de DGE: Metadados
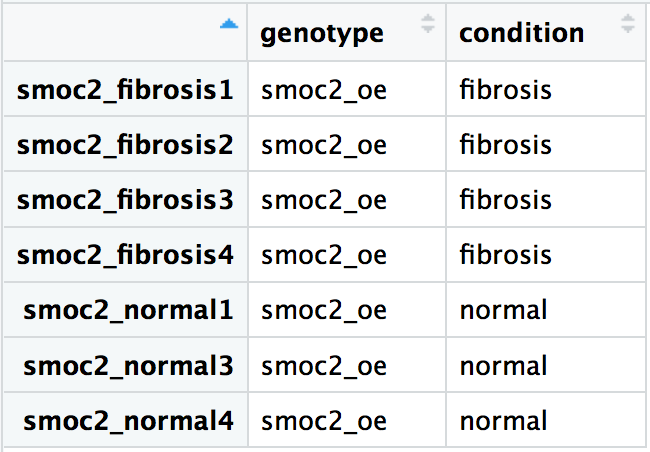
Use as informações abaixo para criar um data frame de metadados para os dados de contagem de fibrose chamado metadata com as colunas genotype e condition. Os nomes das amostras (por exemplo, smoc2_fibrosis1, smoc2_fibrosis2, etc.) devem ser os nomes das linhas do data frame:
Este exercício faz parte do curso
RNA-Seq com Bioconductor em R
Instruções do exercício
- Crie um vetor de caracteres chamado
genotypepara os dados acima usandoc(). - Crie um vetor de caracteres chamado
conditionpara os dados acima usandoc(). - Crie um data frame chamado
smoc2_metadatausandodata.frame()e os vetores de caracteresgenotypeecondition. - Crie um vetor com os nomes das amostras usando
c()e atribua-o aos nomes das linhas do data frame usandorownames().
Exercício interativo prático
Experimente este exercício completando este código de exemplo.
# Create genotype vector
genotype <- c(___)
# Create condition vector
condition <- c(___)
# Create data frame
smoc2_metadata <- data.frame(___, ___)
# Assign the row names of the data frame
rownames(smoc2_metadata) <- c(___)