Teoría de DGE: Metadatos
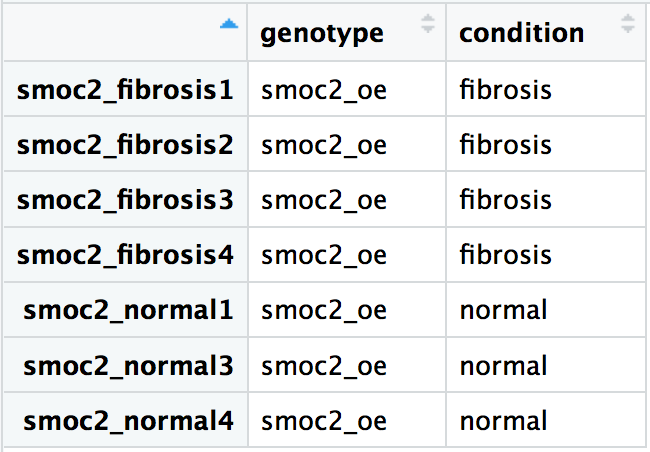
Usa la información de abajo para crear un data frame de metadatos para los recuentos de fibrosis llamado metadata con las columnas genotype y condition. Los nombres de las muestras (p. ej., smoc2_fibrosis1, smoc2_fibrosis2, etc.) deben ser los nombres de fila del data frame:
Este ejercicio forma parte del curso
RNA-Seq con Bioconductor en R
Instrucciones del ejercicio
- Crea un vector de caracteres llamado
genotypepara los datos anteriores usandoc(). - Crea un vector de caracteres llamado
conditionpara los datos anteriores usandoc(). - Crea un data frame llamado
smoc2_metadatausandodata.frame()y los vectores de caracteresgenotypeycondition. - Crea un vector con los nombres de las muestras usando
c()y asígnalo a los nombres de fila del data frame conrownames().
Ejercicio interactivo práctico
Prueba este ejercicio y completa el código de muestra.
# Create genotype vector
genotype <- c(___)
# Create condition vector
condition <- c(___)
# Create data frame
smoc2_metadata <- data.frame(___, ___)
# Assign the row names of the data frame
rownames(smoc2_metadata) <- c(___)