Deduzir MNAR
No exercício anterior, você trabalhou na identificação do tipo de valores ausentes a partir do resumo de ausências. Neste exercício, você vai continuar nessa linha para identificar de forma assertiva dados Missing Not at Random (MNAR).
O resumo de ausências do DataFrame diabetes é o seguinte.
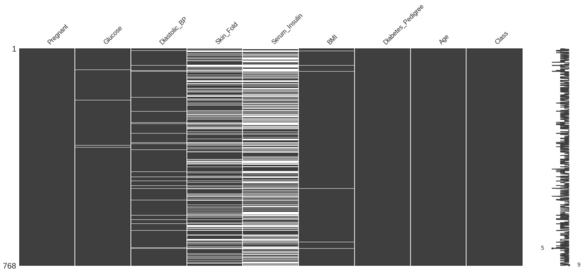
Seu objetivo é ordenar o DataFrame diabetes pela coluna Serum_Insulin e identificar a correlação entre Skin_Fold e Serum_Insulin.
Observe que usamos uma função proprietária display() em vez de plt.show() para facilitar a visualização da saída.
Este exercício faz parte do curso
Lidando com Dados Ausentes em Python
Instruções do exercício
- Importe o pacote
missingnocomomsno. - Ordene os valores da coluna
Serum_Insulinemdiabetes. - Visualize o resumo de ausências de
Serum_Insulincommsno.matrix().
Exercício interativo prático
Experimente este exercício completando este código de exemplo.
# Import missingno as msno
___
# Sort diabetes dataframe on 'Serum Insulin'
sorted_values = ___.___(___)
# Visualize the missingness summary of sorted
___.___(___)
# Display nullity matrix
display("/usr/local/share/datasets/matrix_sorted.png")