Déduire MNAR
Dans l’exercice précédent, vous avez travaillé à identifier le type de valeurs manquantes à partir du récapitulatif de la complétude. Dans cet exercice, vous allez poursuivre sur cette lancée pour identifier de manière certaine des données Missing Not at Random (MNAR).
Le récapitulatif des valeurs manquantes pour le DataFrame diabetes est présenté ci-dessous.
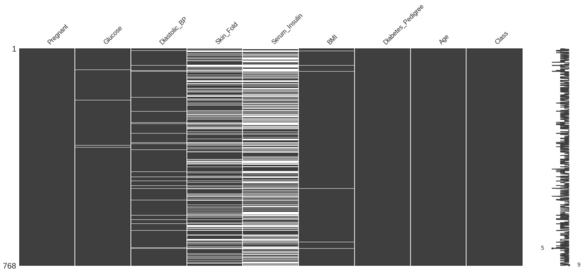
Votre objectif est de trier le DataFrame diabetes selon Serum_Insulin et d’identifier la corrélation entre Skin_Fold et Serum_Insulin.
Notez que nous utilisons une fonction propriétaire display() au lieu de plt.show() afin de vous faciliter la visualisation du résultat.
Cet exercice fait partie du cours
Gérer les données manquantes en Python
Instructions
- Importez le paquet
missingnosous le nommsno. - Triez les valeurs de la colonne
Serum_Insulindansdiabetes. - Visualisez le récapitulatif des valeurs manquantes de
Serum_Insulinavecmsno.matrix().
Exercice interactif pratique
Essayez cet exercice en complétant cet exemple de code.
# Import missingno as msno
___
# Sort diabetes dataframe on 'Serum Insulin'
sorted_values = ___.___(___)
# Visualize the missingness summary of sorted
___.___(___)
# Display nullity matrix
display("/usr/local/share/datasets/matrix_sorted.png")