Gradient Boosted Trees: Visualisierung
Jetzt, da du Modellvorhersagen hast, fragst du dich vielleicht: „Taugen die etwas?“ Es gibt viele Diagramme, mit denen du die Genauigkeit deiner Vorhersagen prüfen kannst; hier schaust du dir zwei gängige an. Erstens ist es hilfreich, ein Streudiagramm der vorhergesagten gegenüber den tatsächlichen Werten zu zeichnen, um den Vergleich zu sehen. Zweitens sollten die Residuen annähernd normalverteilt sein, daher ist ein Dichtediagramm der Residuen nützlich. Die Diagramme sehen in etwa so aus.
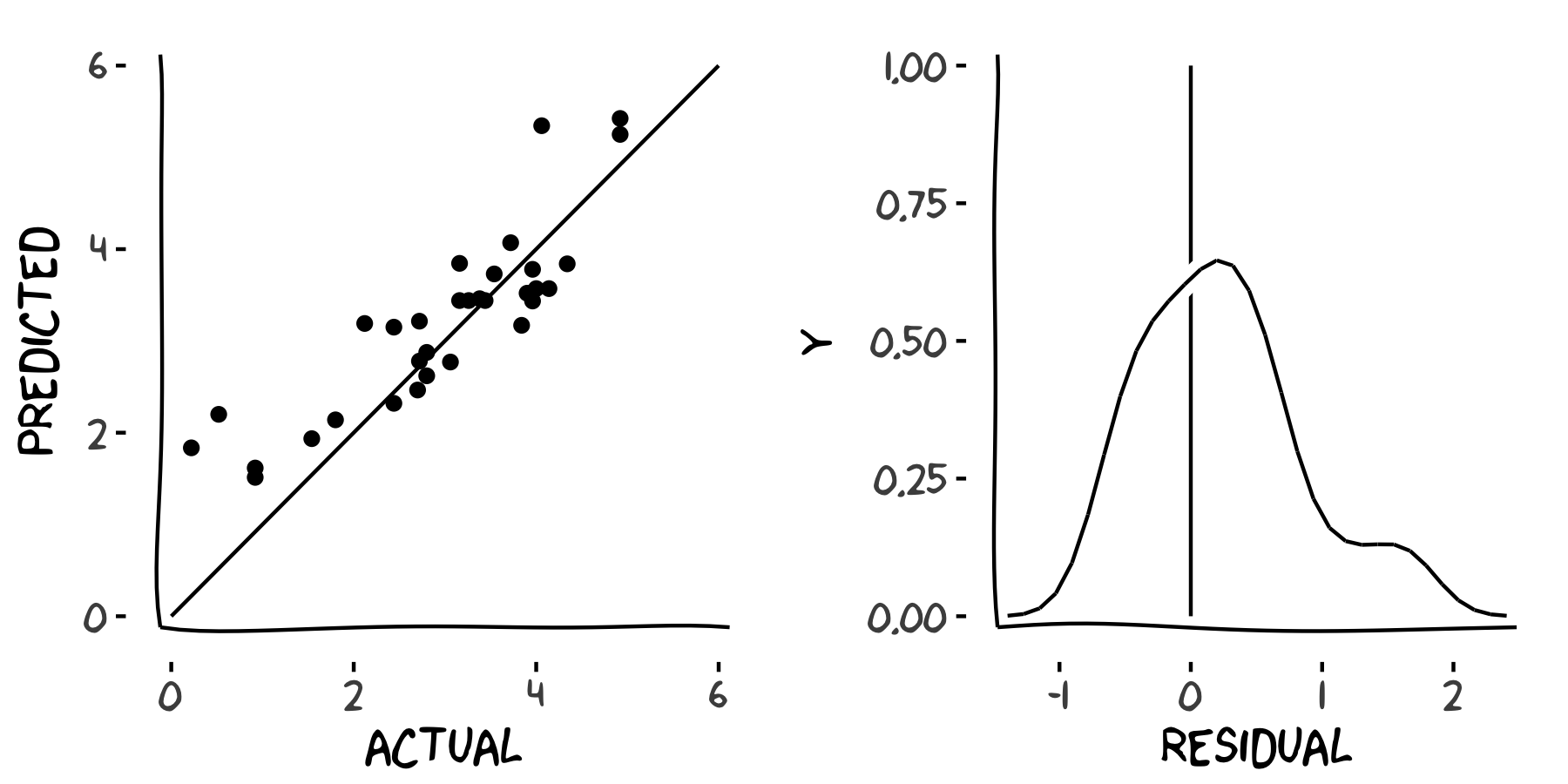
In dieser Übung lernst du, die Residuen selbst zu berechnen (vorhergesagte Werte minus tatsächliche Werte) für deine Modellvorhersagen.
Diese Übung ist Teil des Kurses
Einführung in Spark mit sparklyr in R
Anleitung zur Übung
Ein lokales Tibble responses mit vorhergesagten und tatsächlichen Jahren ist vordefiniert.
- Zeichne ein Streudiagramm „vorhergesagt vs. tatsächlich“.
- Rufe
ggplot()auf. - Das erste Argument ist der Datensatz,
responses. - Das zweite Argument sollte die unzitierten Spaltennamen für x- und y-Achse enthalten (
actualbzw.predicted), verpackt inaes(). - Füge Punkte hinzu, indem du
geom_point()ergänzt. - Mache die Punkte teilweise transparent mit
alpha = 0.1. - Füge eine Referenzlinie hinzu mit
geom_abline()und den Argumentenintercept = 0undslope = 1.
- Rufe
- Erstelle ein Tibble der Residuen namens
residuals.- Rufe
transmute()aufresponsesauf. - Die neue Spalte soll
residualheißen. residualsoll dem vorhergesagten minus dem tatsächlichen Wert entsprechen.
- Rufe
- Zeichne ein Dichtediagramm der Residuen.
- Pipe das transmutierte Tibble zu
ggplot(). ggplot()benötigt eine einzelne Ästhetik,residual, verpackt inaes().- Füge eine Dichtekurve hinzu, indem du
geom_density()aufrufst. - Füge eine vertikale Referenzlinie bei Null hinzu, indem du
geom_vline()mitxintercept = 0aufrufst.
- Pipe das transmutierte Tibble zu
Interaktive Übung
Vervollständige den Beispielcode, um diese Übung erfolgreich abzuschließen.
# responses has been pre-defined
responses
# Draw a scatterplot of predicted vs. actual
ggplot(___, aes(___, ___)) +
# Add the points
___ +
# Add a line at actual = predicted
___
residuals <- responses %>%
# Transmute response data to residuals
___
# Draw a density plot of residuals
ggplot(___, aes(___)) +
# Add a density curve
___ +
# Add a vertical line through zero
___